Clonación del gen de la polifenol oxidasa de aguacate
Este trabajo se basa en una propuesta de una serie de alumnos de cuarto de bioquímica de hace varios años, la cual es claramente una aplicación de los conocimientos de bioquímica adquiridos a lo largo de los años, si bien, el autor de esta entrada de encuentra a penas terminando segundo.
En primer lugar, es necesario contextualizar, exponer la problemática y qué solución se propone, para así entender por qué seguimos los pasos que vamos a mostrar. Esta enzima, la polifenol oxidasa, es una proteína muy extendida en el reino vegetal que se encuentra principalmente en los frutos. La misma y una vez abierto, en este casi el aguacate lleva al pardeamiento típico que observamos, de modo que si logramos inhibir su actividad, lograremos que el aguacate dure más tiempo y por lo tanto, gana interés desde el punto de vista comercial. En este caso, la idea de la que partimos es la inhibición no de la propia enzima, sino de su síntesis por parte del ribosoma, para lo cual aprovecharemos los propios mecanismos de regulación postranscripcional que las células eucariotas poseen, con el empleo de ARN pequeños, en este caso siRNA, ya que son de síntesis exógena. Estos se unen al ARNm de cierta proteína y por complementariedad, total o parcial, bloquean la síntesis de proteínas, por lo que así no habrá expresión de la polifenol oxidasa.
1.Secuencia del gen de la polifenol oxidasa
Es el primer paso, conocer la secuencia que queremos silenciar para así poder diseñar los siRNA. Para ello nos iremos al NCBI, en concreto de la bases de datos de nucleótidos, si bien, nuestra sorpresa es que no se encuentra ninguna polifenol oxidasa de aguacate, de modo que tendremos que hacer una búsqueda de otra forma que hasta ahora no habíamos empleado. Para ello, lo que haremos es buscar el gen de una polifenol oxidasa vegetal, la cual nos sirva como referente a la hora de buscar en el genoma del aguacate, ya que posiblemente la enzima esté conservada. En este caso empleamos el de Malus domestica, o manzana, la cual si se encuentra en la base de datos, la cual tiene como número de acceso D87669.1 y cuya entrada vemos a continuación.
Para seguir, lo que haremos es un BLASTn, donde le pediremos que compare con la secuencia de ADN del aguacate o Persea americana, tal y como se muestra en la siguiente imagen, de modo que podamos localizar en el genoma del mismo al región que posiblemente codifique para la enzima que estamos buscando.
De todas ellas, seleccionamos la primera, por presentar mejores valores estadísticos, lo cual aporta fiabilidad. El resultado del alineamiento se puede ver en la captura que se puede ver bajo estas líneas.
Por tanto, ya tenemos la secuencia del gen a silenciar, de todo el genoma del aguacate, es justo este punto el que nos interesa que no se exprese gracias a la técnica que se ha expuesto al comienzo de este trabajo.
2.Amplificación por PCR
El protocolo ya lo estudiamos en la primera práctica del curso, de modo que solo nos centraremos, de forma muy superficial en el diseño de los primers gracias a la herramienta pick primers que nos facilita el NCBI. Seguiremos el mismo proceso que en aquella vez, con los siguientes ajustes:
Con estos el resultado que se proporciona es el que se muestra abajo:
Seleccionamos la pareja nº2 .
3.Diseño del siRNA
Es el siguiente paso, diseñar las secuencias de ARN de silenciamiento que emplearemos en este trabajo. Para ello empleamos una web (OligoWalk) que nos facilita la secuencia ya demás, la idoneidad de cada una, de modo que elegimos el primero de a imagen que a continuación se muestra.
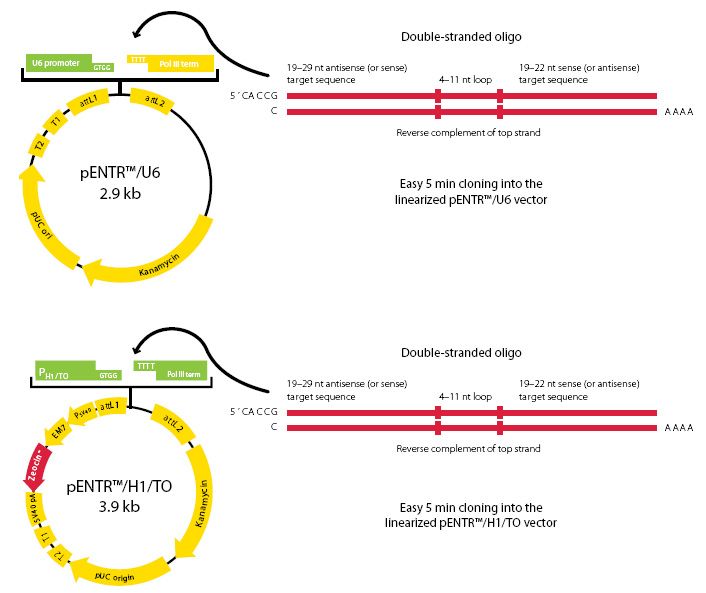
Además de la secuencia hay que introducirlo en la célula, para lo que se usan una serie de vectores, como el que nos facilita la empresa ThermoFisher y cuyo esquema se muestra a continuación. Este vector, intriducido en la célula permite que al expresarse, el ARN que buscamos se sintetice de forma semejante a como lo hace en el mecanismo biológico de regulación del que participa y bloqueará la traducción del ARNm del gen de la polifenol oxidasa.
4.Clonación molecular
Ahora llega el momento de la clonación del gen de la polifenol oxidasa, de modo que debemos insertar el amplicón de la PCR. Como es un gen de eucariota debe expresarse en células eucariotas y someterse a un splicing, de modo que presentamos el siguiente vector para eucariotas:
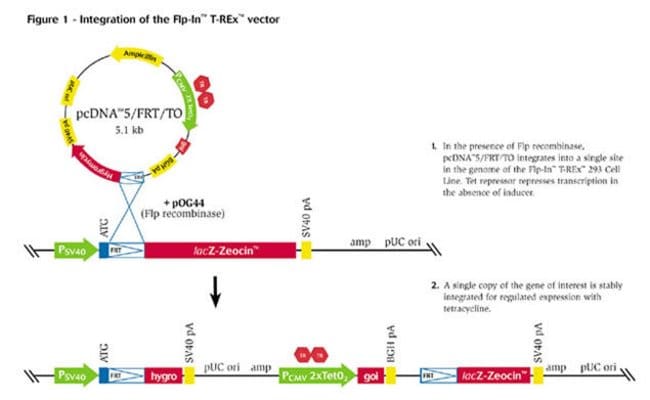
Mediante un protocolo de transformación, tenemos que seguir el protocolo que se adjunta (protocolo del vector de expresión). En concreto en este caso, tendremos que emplear un medio con ampicilina y X-gal además de IGTP (un intensificador de la expresión del gen LacZ). Las colonias que sean blancas son las que nos interesan a nosotros, y son de las que replicaremos para obtener un cultivo transformado.
5.Silenciamiento
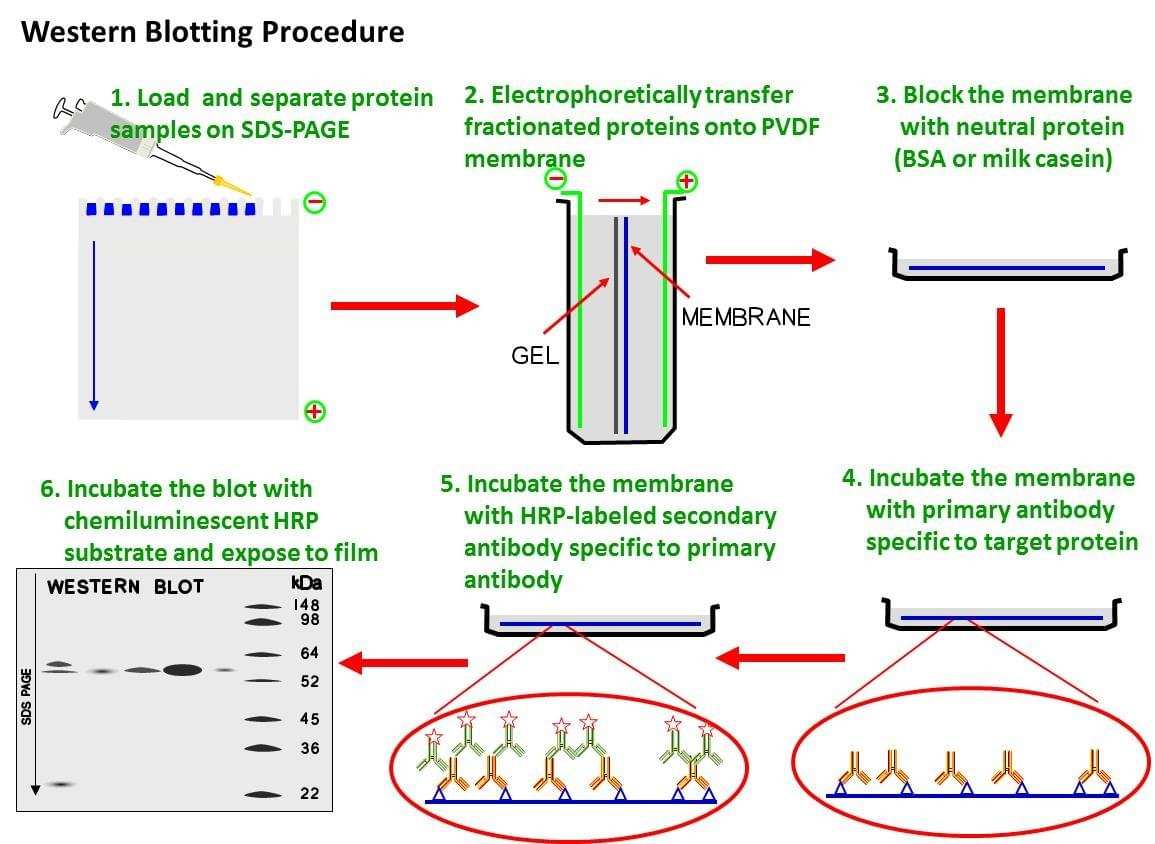
Para ello, transformaremos estas células con el vector de silenciamiento que ya explicamos antes, de modo que, podremso evaluar por blotting la expresión de los genes, gracias a una sonda monolocus, de modo que se hace un proceso parecido al anterior, se toman las colonias transformadas y de estas purificaremos el ARN presente y por un Nothern, hibridaremos con la sonda que diseñaremos y así comprobamos que hay ARNm de la enzima. Posteriormente, por un Western (necesitamos un anticuerpo anti-PPO) podemos evaluar al expresión ( no esperando que haya enzima).
-Nothern blot: se resume en la imagen siguiente.

Primero hay que aislar el ARN de las células (con plásmido del gen y de las silenciadas) para lo que se presenta el siguiente kit de extracción de la empresa Thermofisher del cual se presenta además el siguiente protocolo. Ahora podremos hacer el Nothern, para lo cual hemos encontrado otro kit de la misma empresa y su correspondiente protocolo. En lo que respecta a la sonda, no he logrado encontrar ningún protocolo de síntesis, para l que se me ocurre encargar un oligo o cadena de ADN marcado ya sea por fluorescencia directa o indirecta, del gen que estamos buscando o un fragmento de este, de modo que así hibride con el ARNm y lo detectemos.
Con esto comprobamos la expresión del ARNm en las células sin el vector de silenciamiento, ya que en las silenciadas, habría degradación del ARNm por lo que no se podría detectar, mientras que en las no silenciadas, debe detectarse en la población.
-Western blot: nos servirá como comprobación de los resultados del Nothern Blot anterior.
Se necesita en primer lugar el anticuerpo. En esta web se podría encargar. Después habría que hacer el inmunoensayo, que se resume a continuación:
A la hora de hacer el ensayo, es necesario extraer proteínas, para lo se aporta el siguiente kit y su correspondiente protocolo. Ahora podemos hacer la electroforesis y el inmunoensayo, gracias al siguiente kit siguiendo los pasos que se indican en su protocolo.
El resultado que esperamos es que en las células no silenciadas sí haya detección de la proteína y en las silenciadas que no haya ninguna señal propia de la proteína, ya que eso significa que efectivamente, no se está expresando y que el ensayo ha sido un éxito.
6.Mapa de restricción
Finalmente, se aportan una serie de herramienrtas para diseñar el mapa de restricción del plásmido, lo cual supone saber también si estaba o no insertado el gen de la enzima antes de la transformación de las células. Por ejemplo, el de Biomodel.
CONCLUSIONES
En conjunto, las prácticas del curso y en concreto esta, han supuesto todo un resto, del cual nunca pensé que pudiera salir, ya que no estamos hechos a tener que diseñar por nuestra cuenta una estrategia, sino a seguir paso a paso los protocolos que se nos dan en el laboratorio de prácticas, si bien es normal ya que primero hay que aprender las técnicas y a hacerlas para después poder saber realmente de qué hablamos, ya que el hecho de que casi no haya ido al laboratorio influye, inevitablemente en como enfoco estos trabajos, ya que desconozco trucos o fallos típicos que se aprenden de la experiencia que algún día espero ganar en este ámbito, ya que la bioquímica no es solo teoría, fórmulas y genes, es también manejo del laboratorio, de técnicas y de aplicación de la teoría.